Pendahuluan
Keanekaragaman mikroorganisme merupakan indikator kunci kesehatan ekosistem (Hughes et al., 2001). Namun, pengukuran biodiversitas mikroorganisme secara kuantitatif memerlukan pendekatan yang menggabungkan teknik molekuler dan analisis ekologi.PCR-RFLP (Polymerase Chain Reaction-Restriction Fragment Length Polymorphism) menjadi salah satu metode andalan untuk memprofil komunitas mikroba, sementaraindeks Shannon (Shannon Diversity Index) memberikan kerangka matematis untuk mengkuantifikasi keragaman taksonomi. Artikel ini menjelaskan integrasi kedua pendekatan tersebut dalam studi biodiversitas mikroba.
PCR-RFLP: Dari Amplifikasi DNA ke Profil Komunitas
- Amplifikasi Gen Target:
Gen penanda taksonomi (misalnya 16S rRNA pada bakteri) diamplifikasi menggunakan PCR dengan primer spesifik. Daerah hipervariabel seperti V3-V4 dipilih karena resolusi tingginya untuk membedakan spesies (Weisburg et al., 1991). - Pemotongan dengan Enzim Restriksi:
Produk PCR dipotong oleh enzim restriksi (contoh:HaeIII,MspI) yang mengenali sekuens spesifik. Pola pemotongan menghasilkan fragmen DNA unik untuk setiap kelompok mikroba (OTU –Operational Taxonomic Unit). - Visualisasi Profil:
Fragmen DNA dipisahkan melalui elektroforesis gel agarosa. Setiap pita (band) merepresentasikan OTU, dengan intensitas pita yang berkorelasi dengan kelimpahan relatif (meski perlu hati-hati karena bias PCR) (Schloss et al., 2009).
Indeks Shannon: Kuantifikasi Keragaman Komunitas
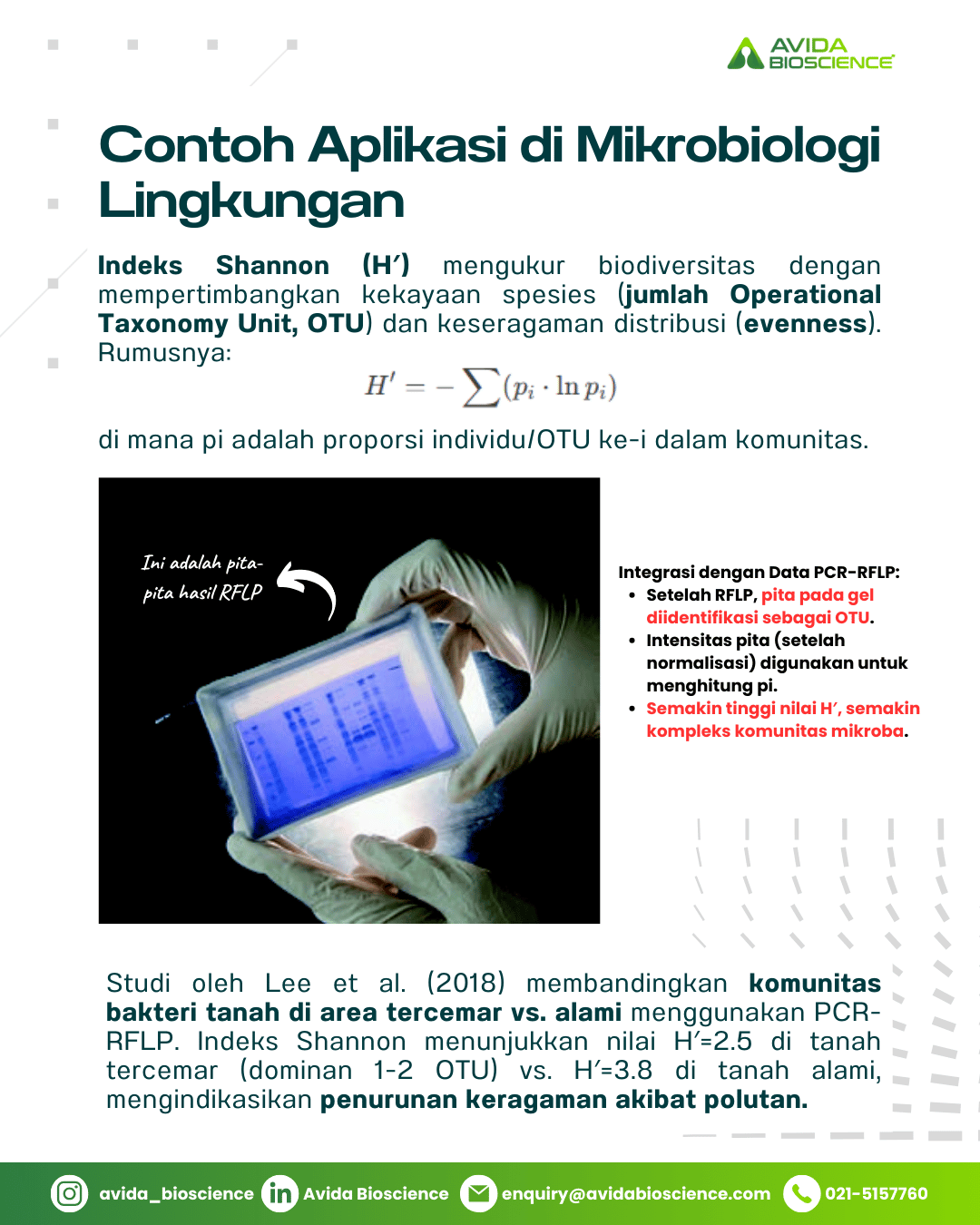
Indeks Shannon (H′) mengukur biodiversitas dengan mempertimbangkankekayaan spesies (jumlah OTU) dankeseragaman distribusi (evenness). Rumusnya:
H′=−∑(pi⋅lnpi)
di manapi adalah proporsi individu/OTU ke-idalam komunitas.
Integrasi dengan Data PCR-RFLP:
- Setelah RFLP, pita pada gel diidentifikasi sebagai OTU.
- Intensitas pita (setelah normalisasi) digunakan untuk menghitungpi.
- Semakin tinggi nilaiH′, semakin kompleks komunitas mikroba.
Contoh Aplikasi:
Studi oleh Lee et al. (2018) membandingkan komunitas bakteri tanah di area tercemar vs. alami menggunakan PCR-RFLP. Indeks Shannon menunjukkan nilaiH′=2.5di tanah tercemar (dominan 1-2 OTU) vs.H′=3.8 di tanah alami, mengindikasikan penurunan keragaman akibat polutan.
Keunggulan dan Tantangan
- Kelebihan:
- Kombinasi PCR-RFLP dan indeks Shannonhemat biaya dan cocok untuk studi komparatif skala kecil.
- Indeks Shannon mudah dihitung menggunakan software sepertiPAST atau R (Hammer et al., 2001).
- Keterbatasan:
- Resolusi RFLP terbatas: OTU mungkin mewakili beberapa spesies yang memiliki situs restriksi serupa.
- Intensitas pita tidak selalu linier dengan kelimpahan seluler karena bias PCR (Pinto & Raskin, 2012).
Studi Kasus: Mikrobioma Usus dan Penyakit Autoimun
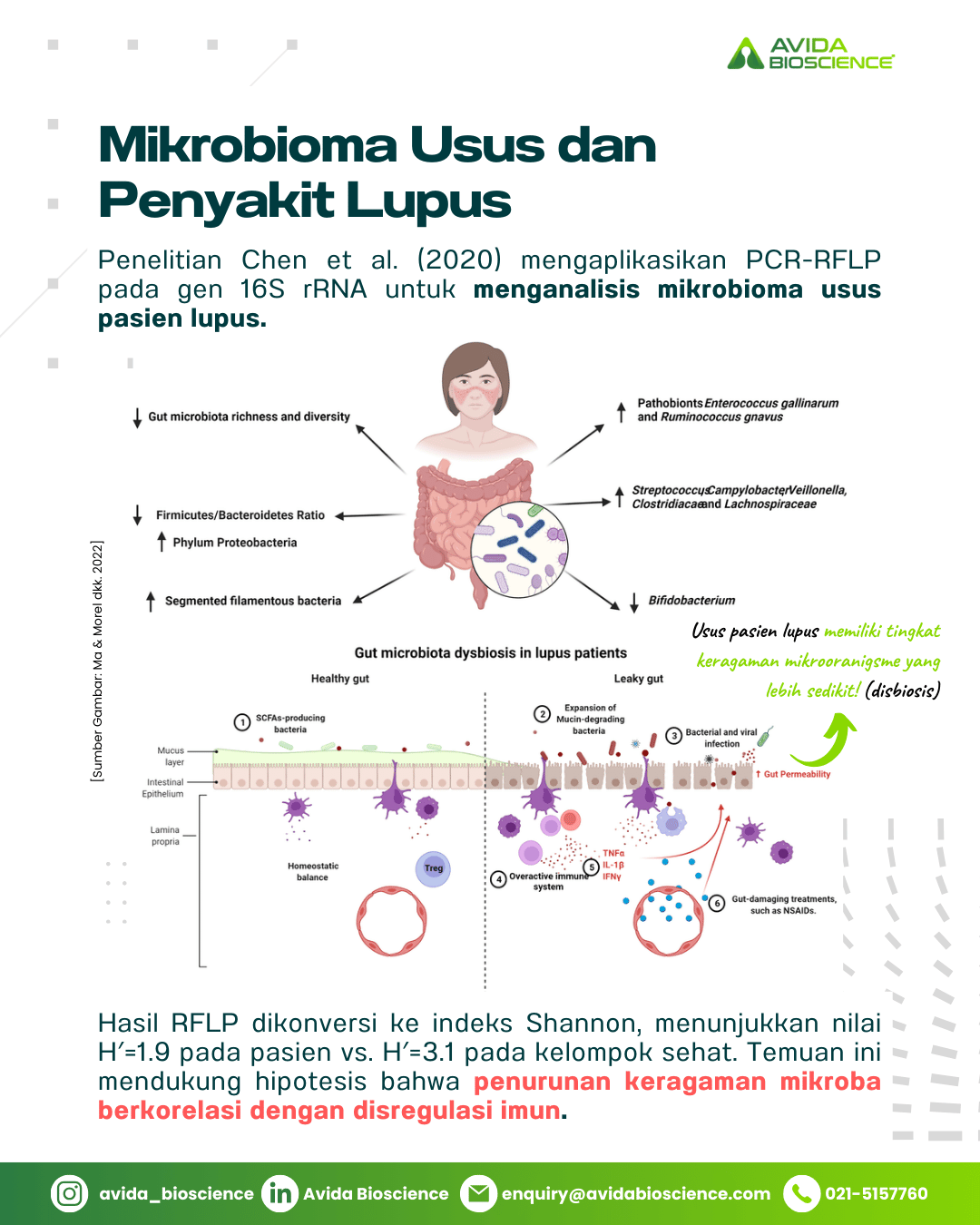
Penelitian Chen et al. (2020) mengaplikasikan PCR-RFLP pada gen 16S rRNA untuk menganalisis mikrobioma usus pasien lupus. Hasil RFLP dikonversi ke indeks Shannon, menunjukkan nilaiH′=1.9pada pasien vs.H′=3.1pada kelompok sehat. Temuan ini mendukung hipotesis bahwa penurunan keragaman mikroba berkorelasi dengan disregulasi imun.
Masa Depan PCR-RFLP di Era NGS
MeskipunNext-Generation Sequencing (NGS) menawarkan resolusi tinggi, kombinasi PCR-RFLP dan indeks Shannon tetap relevan untuk:
- Studi pendahuluan yang memerlukan analisis cepat dan murah.
- Pengukuran biodiversitas di wilayah dengan sumber daya terbatas.
- Validasi data NGS melalui pendekatan komplementer.
Kesimpulan
Integrasi PCR-RFLP dengan indeks Shannon memungkinkan peneliti mengukur biodiversitas mikroba secara kuantitatif tanpa investasi besar. Meski memiliki keterbatasan resolusi, pendekatan ini menjadi jembatan antara teknik konvensional dan analisishigh-throughput.
Referensi
- Chen, Y. et al. (2020). Gut microbiota dysbiosis in systemic lupus erythematosus.Front. Immunol., 11, 1021.
- Hammer, Ø. et al. (2001). PAST: Paleontological statistics software package.Palaeontol. Electron., 4(1), 1-9.
- Hughes, J. B. et al. (2001). Counting the uncountable: Statistical approaches to estimating microbial diversity.Appl. Environ. Microbiol., 67(10), 4399-4406.
- Lee, S. H. et al. (2018). Soil microbial diversity in heavy metal-polluted environments.Environ. Pollut., 241, 1212-1221.
- Pinto, A. J., & Raskin, L. (2012). PCR biases distort bacterial and archaeal community structure in pyrosequencing datasets.PLoS ONE, 7(8), e43093.
- Schloss, P. D. et al. (2009). Introducing mothur: Open-source, platform-independent, community-supported software for describing and comparing microbial communities.Appl. Environ. Microbiol., 75(23), 7537-7541.
- Weisburg, W. G. et al. (1991). 16S ribosomal DNA amplification for phylogenetic study.J. Bacteriol., 173(2), 697-703.
Untuk informasi lebih lanjut, hubungi tim kami di AVIDA Bioscience!